O conteúdo de DNA das bactérias geralmente está dividido em dois tipos de estruturas, o cromossomo bacteriano ou nucleóide e os plasmídeos.
Essas estruturas não são formadas apenas por DNA, existe associação com proteínas (que garantem dobramento e níveis de condensação adequados). Quando comparadas com as interações DNA-proteínas que existem nos cromossomos eucariontes, o dobramento do cromossomo bacteriano é mais simples.
Cromossomo Bacteriano
1) Contem os genes que codificam para proteínas essenciais para o funcionamento da célula.
2) Geralmente é uma estrutura única e circular, formada por uma molécula de DNA que fica presa á parte interna da membrana plasmática (o mesossomo);
3) Possui poucas regiões que não são genes (poucas regiões não codificantes);
4) É comum que genes relacionados com a mesma função ou via metabólica fiquem agrupados e sejam transcritos juntos (em uma mesma fita de mRNA) constituindo uma "unidade de transcrição". A tradução desse mRNA origina as proteínas codificadas pelos genes que fazem parte da região que foi transcrita.
5) Não contém genes interrompidos (são poucas exceções os genes com íntrons)
Plasmídeos
1) Moléculas de DNA circulares e pequenas que ficam livres no citoplasma;
2) Contêm genes que codificam para proteínas relacionadas com vantagens adaptativas como resistência a antibióticos e capacidade de degradar moléculas que eventualmente apareçam no meio;
3) Geralmente cada célula tem vários plasmídeos (iguais ou diferentes);
4) Podem ser transferidos horizontalmente (sem ser por reprodução).
OBS.: A aquisição de plasmídeos depositados no ambiente após a morte de uma célula é um dos mecanismos relacionados com o rápido desenvolvimento de bactérias com resistências múltiplas a antibióticos. Essa aquisição (denominada de transformação) não respeita barreiras entre espécies (plasmídeos de uma espécie podem ser incorporados ao citoplasma de outra espécie).
7) Fimbrias ou Pili e Flagelos
As fímbrias, também chamadas de pili, são composições formadas por filamentos, onde estão ligadas à função de aderência, e conjugação, pois transportam material genético. Os flagelos são composições filiformes utilizados na mobilidade da bactéria. Estão presos na membrana plasmática pelo corpo basal.
As bactérias decompositoras não decompõem somente corpos ou carcaças de organismos vivos, mas também dejetos e secreções como urina, fezes, matéria orgânica em geral, são processados por bactérias. Estes organismos degradam a matéria orgânica em moléculas simples que são liberadas no ambiente e podem ser novamente utilizadas por outros seres vivos. Assimi, elas atuam na reciclagem de nutrientes importantes para outros organismos.
As bactérias de tipo selvagem são prototróficas, i.e., conseguem sintetizar tudo de que necessitam para se multiplicar e se reproduzir quando têm uma fonte de energia e algumas moléculas inorgânicas.
As bactérias mutantes auxotróficas necessitam de outros metabólitos para seu desenvolvimento.
NUTRIÇÃO
SAPRÓFITAS
DECOMPOSITORAS
FOTOSSINTETIZANTES
Quando pensamos em fotossíntese, logo lembramos que um dos seus produtos é o oxigênio, certo? Afinal, sem o processo fotossintético, o oxigênio não completaria seu ciclo voltando para a atmosfera e, nós e os outros seres aeróbios, morreríamos sufocados. (
blogdoenem)
Porém, em algumas espécies de bactérias autótrofas pode ocorrer o processo fotossintético um pouco diferente da fotossíntese que ocorre em cianobactérias e eucariontes, como protistas e plantas. Este processo diferenciado de fotossíntese ocorre em bactérias extremófilas (que vivem em condições extremas em que outros seres vivos não sobreviveriam), como as bactérias verdes sulfurosas e as púrpuras.(
blogdoenem)
Chlorobium tepidum é uma bactéria anaeróbia fototrófica que também é termófila. Como membro da família Chlorobiaceae, é um organismo modelo de bactérias verdes sulfurosas. Os habitats dessa bactéria incluem águas anóxicas e ricas em sulfetoS, lama, sedimentos, esteiras microbianas e até mesmo consórcios microbianos. As células desta bactéria são bastonetes gram-negativos não móveis de comprimentos variados que fotooxidam compostos de enxofre reduzido.
C. tepidum foi isolado de certas fontes termais ácidas de alto sulfeto da Nova Zelândia. Em condições fotoautotróficas, a taxa de crescimento desta bactéria é mais rápida do que qualquer outro fototrófico anaeróbico, pois as gerações são produzidas a cada duas horas. Consequentemente, é um candidato ideal para estudar a fotossíntese e autotrofia em bactérias verdes.
Fig. 1 a, b. Phase contrast photomicrographs of cells of
Chlorobium tepidum strains a TLS and b S. PTE.-I Cultures were grown at 47C for 48 h at 5400 lux. Marker bar = 5 gm. (
WOESE, CASTENHOLZ, MARDIGAN, 1991)
Micrografia eletrônica de varredura (MEV) de Chlorobium tepidum, bactéria Gram-negativa, termofílico, autotrófico obrigatório, sulphur green bacteria. As espécies de Chlorobium crescem em tapetes densos sobre fontes termais e outras águas ricas em sulfeto, lama e sedimentos. C. tepidum é uma bactéria autotrófica que utiliza fotossíntese anoxigênica (não produz oxigênio) e produz enxofre elementar como produto residual. Além disso, pode fotooxidar o hidrogênio, bem como outros compostos de enxofre, como sulfeto, polissulfeto e tiossulfato. C. tepidum tem complexos especiais de coleta de luz chamados clorossomos que contêm bacterioclorofilas e carotenóides. C. tepidum também tem a capacidade de fixar o nitrogênio atmosférico. A utilização de compostos de nitrogênio e enxofre por Chlorobium tepidum também é importante nos ciclos globais de ambos os nutrientes. Ampliação: x2.600 quando o eixo mais curto é impresso em 25 milímetros. (Credit DENNIS KUNKEL MICROSCOPY / SCIENCE PHOTO BIBLIOTECA).
Durante a fotossíntese, estas bactérias não utilizam água, mas sim gás sulfídrico (H2S), liberando enxofre em vez de oxigênio. Lembre-se de que durante a fotossíntese comum, o oxigênio gasoso liberado na fase clara é proveniente da fotólise da água.(
blogdoenem)
Neste caso, como a água não é utilizada como reagente desta reação de fotossíntese, não será liberado oxigênio. Estas bactérias possuem clorofilas especiais chamadas de bacterioclorofilas que possuem composição diferente da clorofila das plantas. Veja a seguir a equação da quimiossíntese, da Fotossíntese bacteriana:
As bacterioclorofilas são pigmentos fotossintéticos que ocorrem em várias bactérias fototróficas. São relacionadas com as clorofilas, pigmentos principais nas plantas, algas e cianobactérias. Os grupos que contêm bacterioclorofila realizam fotossíntese, mas não produzem oxigénio. Usam comprimentos de onda que não são absorvidos em plantas. Diferentes grupos possuem diferentes tipos de bacterioclorofilas:
Bacterioclorofila a Bactérias purpuras
Bacterioclorofila b Bactérias purpuras
Bacterioclorofila c Bactérias verdes sulfurosas, Chloroflexi
Bacterioclorofila d Bactérias verdes sulfurosas
Bacterioclorofila e Bactérias verdes sulfurosas
Bacterioclorofila g Heliobactérias
As bacterioclorofilas c e d são clorinas, com um anel pirrol reduzido, e as outras são bacterioclorinas, com dois anéis.
Chlorobium tepidum, também conhecida como bactéria verde sulfurosa, é uma espécie de bactéria autotrófica, que possui pigmentos verdes (bacterioclorofila C) agregados numa forma espacial diferente das plantas e são necessários para captar a energia solar. Estes agregados são extremamente eficientes na captação e transmissão da energia luminosa.
A sulfobactéria verde é uma bactéria termófila modelo. Estas bactérias usam compostos reduzidos de enxofre, tal como o sulfato, durante a fotossíntese como fornecedor de potencial redutor e não a água.Neste tipo de bactérias o dióxido de carbono não é fixado pela rubisco no ciclo de Calvin que não possuem, mas sim, no ciclo do ácido acético invertido ou ciclo de Krebs invertido.
QUIMIOSSINTETIZANTES
Assim como a fotossíntese, a quimiossíntese é um processo autotrófico de conversão de moléculas de carbono, como dióxido de carbono (CO2) e metano (CH4), em matéria orgânica que será utilizada como alimento pelo organismo.
Porém, no lugar da energia luminosa, as reações que compõem a quimiossíntese utilizam a energia liberada pela oxidação de determinadas moléculas inorgânicas. Esta energia esta armazenada nas ligações covalentes dos compostos que serão oxidados. Essa fonte de energia vem de moléculas contendo elementos como enxofre (S), hidrogênio (H), nitrogênio (N), manganês (Mg) ou ferro (Fe).
Os organismos quimiossintetizantes incluem bactérias e arqueobactérias que vivem em ambientes carentes de luz.
Uma das reações de quimiossíntese mais conhecidas vem de bactérias que utilizam sulfeto de hidrogênio (H2S) como uma fonte de energia, funcionando como base de diversas cadeias alimentares de áreas próximas a fontes termais submarinas. Essas bactérias existem como organismos vivos livres ou formam relações de simbiose com outros organismos dessa região. Um bom exemplo dessas relações é o caso dos poliquetas conhecidos como pogonóforos. Esses animais são anelídeos que formam tubos verticais próximos a fontes termais no fundo dos oceanos e se associam a bactérias quimiossintetizantes, que vivem em um órgão do sistema digestivo do poliqueta. Estas bactérias produzem glicose (C6H12O6) a partir da oxidação do sulfeto de hidrogênio liberado pelas fontes termais, proporcionando assim uma fonte de alimento tanto para si quanto para o hospedeiro pogonóforo. A equação geral do processo de quimiossíntese utilizando sulfeto de hidrogênio é a seguinte:
12 H2S + 6 CO2 = C6H12O6 + 6 H2O + 12 S
Ao observar-se a reação acima, percebrmos a semelhança com a reação que produz glicose através da fotossíntese, exceto o fato que a fotossíntese libera gás oxigênio, enquanto a quimiossíntese produz enxofre sólido. Os grânulos de enxofre amarelo são visíveis no citoplasma de bactérias que realizam a reação.
Além dessa, bactérias que vivem no solo no ambiente terrestre também são capazes de realizar quimiossíntese, sendo mais conhecidas as que utilizam compostos de nitrogênio como fonte de energia. Essas bactérias são chamadas de nitrobactérias e pertencem aos gêneros
Nitrosomonas e
Nitrobacter, sendo essenciais no ciclo do nitrogênio em nosso planeta (
infoescola).
As bactérias do gênero Nitrosomonas oxidam a amônia (NH3) presente no solo, transformando-o em íon nitrito (NO2–) e liberando energia.
Em seguida, essa energia é utilizada para a produção de glicose a partir do gás carbônico dissolvido na àgua. A seguir estão as equações gerais das reações de quimiossíntese realizadas pelas Nitrosomonas:
2 NH3 + 3 O2 = 2 NO2– + 2 H2O + 2H+ + Energia
6 CO2 + 6 H2O + Energia = C6H12O6 + 6 O2
As bactérias do gênero Nitrobacter, por sua vez, oxidam o íon nitrito (NO2–) transformando-o em íon nitrato (NO3–) em uma reação que também libera energia.
Da mesma forma que as Nitrossomonas, as Nitrobacter utilizam essa energia para produzir moléculas orgânicas de glicose a partir do CO2, seguindo as seguintes reações:
2 NO2– + O2 = 2 NO3– + Energia
6 CO2 + 6 H2O + Energia = C6H12O6 + 6 O2
7. Papel das bactérias
Bactérias são igual a doenças? Normalmente pensamos em patogenias quando ouvimos a palavra bactéria. No entanto, bactérias que causam doenças são uma pequena parte das Eubactérias. As bactérias são importantíssimas e essenciais para a manutenção da vida em nosso planeta, uma vez que desempenham o papel de decompositores em todos os ecossistemas, estando portanto na base de todos os ecossistemas existentes.
Foram os primeiros seres vivos a aparecer em nosso planeta por volta de 3,8 bilhões de anos atrás e os primeiros organismos a produzir e liberar oxigênio, reduzindo a concentração de gás carbônico na atmosfera. Um fato inegável é que cloroplastos e mitocôndrias são bactérias que em eventos de endosimbiose "produziram" a célula eucariota ao invadir uma protocélula.
As bactérias tem papel essencial no ambiente como saprófitas (seres vivos que se nutrem de matéria orgânica em decomposição) e decompositores (que promovem ativamente a deconposição da matéria orgânica; ambas promovendo a ciclagem dos nutrientes (reciclagem da matéria orgânica). Com isso, os nutrientes como N2, CO2, Po4, K, Ca, Zn etc, voltam para os seu respectivos ciclos biogeoquímicos, possibilitando seu uso por outros organismos.
Ciclo do carbono
Ciclo do Nitrogênio
O Nitrogênio é um elemento químico com símbolo N, número atómico 7 e de massa atómica 14,00674 u (7 prótons e 7 neutrons, com adição da pequena massa dos 7 elétrons), representado no grupo 7 da tabela periódica. Esse gás foi descoberto pelo médico escocês Daniel Rutherford em 1772, como componente separável do ar. Em condições normais forma um gás diatómico (N2), incolor, inodoro, insípido e principalmente inerte, não participando da combustão e nem da respiração, todavia pode formar bolhas no sangue sob pressão intensa (volta a forma de gás). O grupo do nitrogênio é o décimo quinto (15) grupo da tabela periódica, como todos os elementos dessa família, possui 5 elétrons na camada de valência. Eles são ocasionalmente chamados de pnicogênios ou pnigogênicos, palavra derivadas do grego ( πνῑ́γω, pnigo: sufocar; e Γέν, gén: generar ) que significam "sufocar". No grupo há uma transição clara do caráter não metálico para metálico quando se percorre a família de cima para baixo.
Complete a sua tabela periódica
O nitrogênio é indispensável à vida, uma vez que entra na constituição dos aminoácidos, proteínas, nas bases nitrogenadas que constituem os ácidos nucléicos.
Admite-se, que o corpo humano seja constituído de 16% de proteínas. A mais importante fonte de nitrogênio para todos os seres vivos é a atmosfera.
O nitrogênio constitui cerca de 75 a 78% do volume do ar atmosférico, na forma de N2. Todavia, a maioria dos
seres vivos é incapaz de aproveitá-lo, em seu metabolismo, na forma em que se encontra na atmosfera; necessitando de um intermediário para fornecer esse gás na forma que possa ser absorvido e usados pelos tecidos vivos. O N2 atmosférico necessita ser fixado na forma de NH3, NO2, NO3 para que possa ser absorvido pelas raízes das plantas e então disponibilizados para os herbívoros e no corpo destes para os carnívoros. Assim, no ciclo do nitrogênio participam bactérias fixadoras, algas verde-azuladas (cianofíceas) e
fungos.
Mesmo que todos os organismos possuam nitrogênio em seus tecidos e necessitem desse elemento químico, nenhum ser vivo excetuando as Eubactérias são capazes de usá-lo na forma em que se encontra na atmosfera, ou seja em sua forma molecular N2.
Algumas espécies de bactérias são capazes de absorver o N2 e utilizá-lo como fonte de energia, incorporando os átomos de nitrogênio em suas moléculas orgânicas e disponibilizando-o em outras formas moleculares para o uso de diversas espécies. A esse processo de incorporação de nitrogênio em moléculas orgânicas a partir do N2 dá-se o nome de fixação do nitrogênio, sendo uma etapa essencial do ciclo do nitrogênio. Da mesma forma, as bactérias que realizam a fixação são chamadas de fixadoras de nitrogênio.
Elas podem ter vida livre como é o caso das cianobactérias, ou viver em simbiose com outros organismos.

A oxidação microbiana da amônia (ou amônio) via nitrito em nitrato é uma etapa central no ciclo do nitrogênio. Este processo, chamado nitrificação, foi pensado para envolver duas etapas separadas mediadas por microrganismos distintos: a oxidação inicial da amônia a nitrito, que é catalisada por microrganismos oxidantes de amônia (AOMs; incluindo bactérias (AOB) ou arquéias (AOA)); seguido pela oxidação de nitrito a nitrato, que é catalisada por bactérias oxidantes de nitrito (NOB).
Bactérias associadas ao ciclo do nitrogênio (
profpc).
Simbiose entre Rhizobium e plantas
Essa interação constiúi uma interação complexa e específica entre as bactérias do solo pertencentes aos gêneros Rhizobium, Sinorhizobium, Mesorhizobium, Phylorhizobium, Bradyrhizobium e Azorhizobium, comumente conhecidos como rizóbios, e suas plantas hospedeiras resultam no desenvolvimento de nódulos radiculares.
TEM micrograph of three Rhizobium leguminosarum bv. trifolii CC275e cells. The length of the bar = 1 µm. (Delestre et all, 2015).
Imagens de Rhizobium leguminosarum bv. viciae cepa GB30 usando microscopia eletrônica de varredura (esquerda) e transmissão (centro) e a aparência morfológica da colônia em meio sólido ½LA (direita) (Mazur et all, 2015)
Genome sequence of the clover symbiont
Rhizobium leguminosarum bv. trifolii strain
CC27
Nódulos são novos órgãos que consistem principalmente de células infectadas das plantas com bacteróides que provovem a fixação do nitrogênio. Dentro dos nódulos, as bactérias assumem uma forma endosimbiótica, os bacteróides, que são capazes de reduzir o nitrogênio atmosférico à amônia (BROUGHTON et al., 2006).
Como a amônia é tóxica para a planta, ela é rapidamente convertida a amidas e/ou ureídos, que podem ser usados pela planta hospedeira para sua nutrição.
A iniciação da formação dos nódulos é controlada em vários níveis: as raízes das plantas excretam vários flavonóides, alguns dos quais necessários para ativar a expressão de genes reguladores da nodulação, particularmente as proteínas NodD (PERRET et al., 2000). O complexo NodD-flavonoides interage com uma região promotora conservada (nod boxes) que está localizada após a maioria dos genes nod no genoma da bactéria.
Fatores determinantes da nodulação em Rhizobium spp NGR234 induzidos por flavonóides. (A) Flavonóides excretados pelas raizes ativam a expressão de genes do rizóbio requeridos para o processso de nodulação (nod, nol, e noe). (B) Fatores Nod são lipo-quito-oligossacarídeos modificados como os β-1,4-oligomeros conectados de N-acetil-D-glucosamina, com um ácido graxo subtiuindo o grupo N-acetil em suas terminações não redutoras. (BROUGHTON et alii, 2004, citado por Santos & Reis, 2008).
A transcrição desses genes acontece e e o produto enzimático dos genes nod leva a síntese e secreção de fatores Nod (lipo-quito-oligossacarídeos) (BROUGHTON et al., 2006). Um controle fino da transcrição dos genes nod está provavelmente relacionado à variações na seqüência das regiões conservadas nos promotores nod, individualmente (FREIBERG et al., 1997).
Outros produtos do rizóbio são necessários para continuidade do desenvolvimento do cordão de infecção, e estes representam um terceiro conjunto de sinais. Embora seja claro que os fatores Nod permitem ao rizóbio penetrar nas raízes das leguminosas, outros carboidratos bem como proteínas são necessários para o desenvolvimento do cordão de infecção e e as subseqüentes etapas da formação do nódulo nas raízes. (
SANTOS E REIS, 2008).
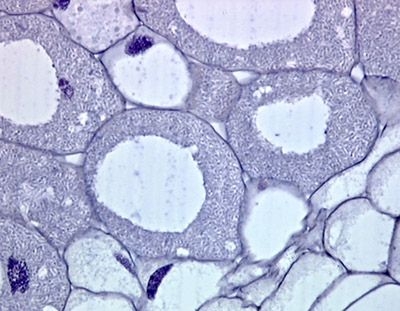
Nesta figura podemos notar dois nódulos coletados em raízes de Favas,
Vicia faba L. 1753, que foram cortados e corados com Cristal violeta,
que como sabemos cora indiscriminadamente todas as bactérias.
Nessas duas figuras acima as células grandes são fortemente invadidas por bactérias do gênero
Rhizobium e às vezes também podem apresentar vacúolos citoplasmáticos grandes. (
UMAR, 2001)
A metade esquerda da imagem acima ilustra essas grandes células preenchidas por bactérias do gênero Rhizobium. Observe que os núcleos também estão aumentados e até mesmo os nucléolos (envolvidos por um halo) são visivelmente proeminentes. No lado direito da imagem, vemos células da plantas vacuoladas e uma ilha microscópica de células de meristema que podem dar origem a um novo ramo de raiz. Esta imagem foi obtida usando uma objetiva de 25x.
Imagem obtida usando uma Objetiva de 40X.
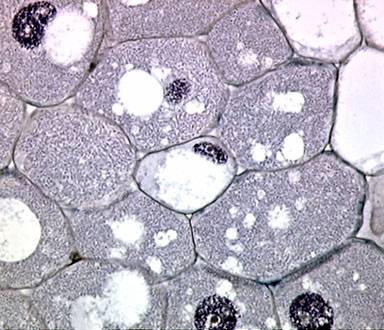
As imagens a seguir correspondem a ampliações maiores dessas células invadidas pela bactéria do gênero Rhizobium. Essa bactéria têm 1 a 2 µm de comprimento e 0,1 µm de diâmetro.
Observe que as bactérias são mostradas nas imagens como uma massa invasiva cinza azulada e não podem ser resolvidas, exceto na maior ampliação. Algumas das células foram coradas apenas com Hematoxilina (uma coloração universalmente reconhecida, bastante específica, para ácido nucleico e núcleos) enquanto outras foram coradas com cristal violeta.
(UMAR, 2001)
Esta imagem foi obtida usando uma objetiva com ampliação de 40X e uma ocular de 1,6 X. As células preenchidas com bactérias possuem um tamanho entre 100 e 200 µm. Células coradas com cristal violeta.(
UMAR, 2001).
Note que todo o citoplasmas de todas as células da planta estão
cheias de bactérias (coradas com uma cor violeta acinzentado).
Células de fava coradas com cristal violeta
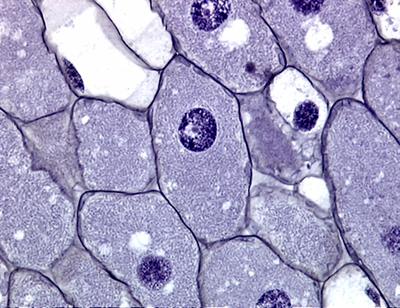
Como podemos observar torna-se claro que essas bactérias do gênero Rhizobium vivem principalmente no interior das células (simbiose intracelular).
Observe que não apenas alguns tipos de bactérias são intracelulares, mas todos os vírus vivem e se replicam dentro das células vivas. Eles são então corretamente chamados de "parasitas intracelulares obrigatórios", como já vimos em nossas aulas de
Virologia.
Elas são capazes de alterar processos metabólicos e moleculares nas células e causar aumento celular. Os nódulos formados por esta proliferação bacteriana existem ao lado e dentro do sistema de raízes em crescimento das plantas hospedeiras. Essas formas tumorais são certamente estruturas benignas e mutuamente úteis, conduzindo a uma maior fixação de N2 e conversão deste em amônia para as plantas hospedeiras. Os nódulos rizobianos são o resultado de uma evolução longa e seletiva, um bom exemplo de convivência com benefícios mútuos.
FIXAÇÃO BIOLÓGICA DO NITROGÊNIO
OU BIOFIXAÇÃO
O nitrogênio está presente em diversas formas na biosfera. A atmosfera contém uma vasta quantidade, cerca de 78% por volume, é composto de nitrogênio molecular (N2). Entretanto, essa grande quantidade não está diretamente disponível para os organismos vivos. A obtenção de nitrogênio da atmosfera requer quebra de uma ligação tripla covalente de excepcional estabilidade, ou seja, é uma ligação de grande energia, entre os dois átomos de nitrogênio (como apresentado na figura abaixo) para produzir amônia ou nitrato. Tais reações, conhecidas como fixação do nitrogênio, podem ser obtidas por processo natura como em relâmpagos e vulcões e industrial com grande gasto de energia e ainda por processo natural ou ação biológica, sem nenhum gasto.
Nitrogênio molecular
O grande reservatório de nitrogênio é a atmosfera terrestre. Embora a atmosfera seja constituída por 78% de nitrogênio (N2) (RAVEN, EVERT E EICHORN, 2007) a maioria dos seres vivos não tem a capacidade de usar diretamente esse elemento da atmosfera para produzir seus aminoácidos e outras substancias orgânicas. Esses organismos são dependentes de compostos nitrogenados mais reativos, como o amônio e o nitrato, presentes no solo. Infelizmente, esses compostos não são tão abundantes como o nitrogênio gasoso. Por isso, embora seja abundante, na atmosfera, a escassez de nitrogênio é frequentemente o principal fator limitante do crescimento das plantas.
O processo pelo qual essa limitada quantidade de nitrogênio circula e recircula por todos os organismos vivos é conhecido como o ciclo do nitrogênio.
(Fonte: Translated and slightly modified by Pedro Spoladore,
based on image by Johann Dréo,
WP)
Ciclo do nitrogênio (Fonte: modif. de ilsabrasil, 2020)
Grande parte do nitrogênio do solo é derivada de organismos mortos e esta sob a forma de materiais orgânicos complexos, como proteínas, aminoácidos, ácidos nucleicos e nucleotídeos. Essas substâncias nitrogenadas são normalmente quebradas rapidamente em compostos simples por bactérias saprófitas e por vários fungos presentes no solo. Esses organismos incorporam o nitrogênio em aminoácidos e proteínas e liberam o excesso sob a forma de íons amônio NH4+ por meio do processo denominado amonificação ou mineralização do nitrogênio. Em meio alcalino, o nitrogênio pode ser convertido em gás amônia (NH3), mas essa conversão ocorre naturalmente apenas durante a decomposição de grandes quantidades de material rico em nitrogênio, como em um monte de esterco ou em uma pilha de compostos ou de resíduos orgânicos em contato com o ar atmosférico. No solo a amônia produzida pelo processo de amonificação é dissolvida na água onde se combina com prótons para formar o íon amônio. Em alguns ecossistemas o íon amônio não é rapidamente oxidado, permanecendo no solo. As plantas que crescem nesse solo são capazes de absorver o NH4+ e usá-lo na síntese de proteína vegetal.
Em alguns solos as bactérias nitrificantes convertem o amônio em nitrito e depois em nitrato
Várias espécies comuns do solo são capazes de oxidar a amônia ou íons amônio. A oxidação da amônia ou nitrificação é um processo que libera energia, e a energia liberada é usada por essas bactérias para reduzir o CO2 (dióxido de carbono), e usá-lo para produzir compostos orgânicos pela bactéria, essas bacaterias são conhecidas como autotróficas quimiossintetizantes.
A bactéria quimiossintetizante e nitrificante Nitosomonas é responsável pela oxidação da amônia a íons nitritro (NO2-):
2NH4+ + 3H2O = 2NO2- + 4H+ + 2H2O
O nitrito é tóxico para as plantas, mas ele raramente se acumula no solo. Nitrobacter, outro gênero de bactérias, oxida o nitrito para formar íons nitrato (NO3+) novamente com liberação de energia:
2NO2- + O2 = 2NO3-
Devido à nitrificação, o nitrato é a forma na qual todo o nitrogênio é absorvido pela maioria das plantas que crescem em terra seca, onde a nitrifacação é é muito favorescida pelas práticas de aração, que oxigenam o solo. A maioria dos fertilizantes nitrogenados usados comercialmente contém tanto íons amônio (NH4+) quanto a uréia, a qual libera NH4+ nos solos. O NH4+ é convertido para NO3- pela nitrificação.
O sistema solo-planta, além de reciclar o nitrogênio, tambem perde esse elemento
A principal perda de nitrogênio no sistema solo-planta ocorre pela desnitrificação, um processo anaeróbico no qual o nitrato é reduzido a formas voláteis de N, como o gás nitrogênio gasoso (N2) e o óxido nitroso (N2O) os quais retornam à atmosfera. Esse processo é executado por numerosos microrganismos. Sabe-se de longa data, que são necessárias condições de baixa concentração de oxigênio para a desnitrificação, característica presente em solos encharcados e outros habitats, como pântanos e brejos. Os pesquisadores agora observaram que esta condição existe dentro dos agregados do solo, mesmo na ausência de água excessiva. Em conseqüência disso a desnitrificação é um processo quase universal nos solos. Um suprimento de material orgânico fresco e pronto para ser decomposto fornece a fonte de energia requerida pelas bactérias desnitrificantes, e, se outras condições são apropriadas, há promoção da desnitrificação. A falta de uma fonte de energia permite o aumento da concentração de nitratos na água do solo até níveis altos. O nitrogênio também é perdido em um ecossistema devido à remoção de plantas (na colheita), à erosão, à destruição da cobertura vegetal pelo foto e à lixiviação. Os nitratos e os nitritos são ânions particularmente susceptíveis de serem retirados da zona radicular pela água que percola o solo.
FIXAÇÃO
A reposição do nitrogênio ocorre principalmente pela sua fixação
Se o nitrogênio que é é removido do solo não fosse constantemente reposto, praticamente toda a vida neste planeta desapareceria aos poucos.
O nitrogênio é reposto no solo principalmente pela sua fixação. Uma quantidade muito menor é adicionada pela precipitação atmosférica (raios de tempestades) e pelo intemperismo de rochas.
A fixação do nitrogênio é o processo pelo qual o N2, atmosférico é reduzido a NH4+ e assim fica disponível para ser transferido para compostos contendo carbono para produzir aminoácidos e outras substâncias orgânicas contendo nitrogênio.
A fixação de nitrogênio, que pode ser executada somente por certas bactérias, é um processo do qual todos os organismos vivos são dependentes, da mesma forma como a maioria dos organismos é dependente da fotossíntese como fonte de sua energia.
A enzima que catalisa a fixação do nitrogênio é chamada nitrogenase. Esta enzima é similar em todos os organismos dos quais foi isolada. A nitrogenase contém grupos prostéticos de molibdênio, ferro e sulfato, e por isso esses elementos são essenciais para a a fixação do nitrogênio.
A nitrogenase também utiliza grandes quantidades de ATP como fonte de energia, tornando a fixação do nitrogênio um processo metabólico caro.
As bactérias fixadoras de nitrogênio podem ser classificadas de acordo com seu modo de nutrição: aquelas que são de vida livre (não-simbióticas) e aquelas que vivem em associação simbiótica com determinadas plantas vasculares.
As bactérias fixadoras de nitrogênio mais eficientes formam associações simbióticas com as plantas
Das duas classes de organismos fixadores de nitrogênio, as bactérias simbióticas constituem, de longe, a mais importante em termos da quantidade total de nitrogênio fixado. As bactérias fixadoras de nitrogênio mais comuns são Rhizobium e Bradyrhizobium, as quais penetram nas raízes de leguminosas como alfafa (Medicago sativa), trevos (Trifolium spp), ervilha (Pisum sativum), soja (Glycine max) e diversos tipos de feijão (Phaseolus spp).
Na associação simbiótica entre as bactérias e as leguminosas, as bactérias suprem a planta com uma forma de nitrogênio que pode ser usada na síntese das proteínas. A planta, por sua vez, supre as bactérias com uma fonte de energia para sua atividade de fixação de nitrogênio e com moléculas que contêm carbono, as quais são necessárias para a produção de compostos nitrogenados.
Os efeitos benéficos no solo promovidos pelo crescimento de plantas leguminosas têm sido observados por séculos. Teofrasto, que viveu no terceiro século a. C., escreveu que os gregos usavam a fava (Vicia faba) para enriquecer os solos.
Na agricultura moderna, é prática comum fazer a rotação de uma planta cultivada não-leguminosa, como por exemplo O milho (Zea mays), e uma leguminosa, como a alfafa, ou milho com a soja ou ainda trigo (Triticum sp). As plantas leguminosas são então colhidas para feno, deixando para trás as raízes ricas em nitrogênio, ou, ainda melhor, as plantas inteiras são simplesmente incorporadas ao solo.
Uma plantação de alfafa que é incorporada de volta ao solo pode adicionar em torno de 300 a 350 quilogramas de nitrogênio por hectare. Em uma estimativa modesta é adicionada à superfície da Terra, a cada ano, 150 a 200 milhões de toneladas de nitrogênio fixado por esses sistemas biológicos.
Nódulos
Nódulos São Produzidos pela Raiz da Planta Hospedeira no Local de Infecção das Bactérias
As bactérias Rhizobium e Bradyrhizobium, comumente chamadas rizóbios, entram nos pêlos radiculares de plantas leguminosas quando estas estão ainda no estágio de plântulas.
O estabelecimento da simbiose entre Bradyrhizobium japonicum e a soja (Glycine max) para a fixação de nitrogênio começa com a ligação dos rizóbios aos pêlos radiculares emergentes, em resposta a atraentes químicos liberados do pêlo radicular (produzidos e liberados pelos pêlos da raiz). Os pêlos radiculares tipicamente transformam-se m estruturas curvadas que aprisionam os rizóbios.
A entrada de rizóbios nos pêlos radiculares e nas células corticais subjacentes ocorre através da formação dos canais de infecção, que são estruturas tubulares formadas pelo crescimento intrusivo e progressivo da parede das células dos pêlos radiculares, a partir do sítio de penetração.
Um único pêlo radicular pode ser penetrado por muitos rizóbios e por isso conter vários canais de infecção. O simbionte bacteriano também induz divisão celular em determinadas regiões do córtex, entrando nessas regiões através do crescimento e da ramificação do canal de infecção. Os rizóbios que estão no canal de infecção são liberados com um envoltório derivado da membrana plasmática da célula hospedeira. Quando os rizóbios aumentam de tamanho e tornam-se realmente fixadores de nitrogênio, são chamados de bacterióides.
A proliferação contínua tanto dos bacterióides como das células corticais da raiz resulta na formação de crescimentos tumorais conhecidos como nódulos. Os processos de infecção e formação do nódulo nas raízes de outras leguminosas são aparentemente similares aos da soja. (RAVEN, EVERT E EICHORN, 2007).
Os nódulos radiculares das leguminosas consistem anatomicamente em um córtex relativamente fino que margeia uma grande zona central, contendo células infectadas ou não por bacterióides. Os fixes vasculares que se estendem a partir do ponto de junção do nódulo com a raiz ocorrem no córtex interno. Na soja, tanto células infectadas quanto não-infectadas participam na produção de ureídeos (derivados de uréia), o produto final da fixação de nitrogênio que é exportado do nódulo para a planta.
Outras leguminosas produzem composts nitrogenados diferentes, sendo um dos mais comuns o aminoácido asparagina. A concentração de O2, nas células infectadas pelos bacterióides deve ser cuidadosamente regulada, já que o O2, é um potente inibidor irreversível da enzima nitrogenase.
Ao mesmo tempo, o O2 (oxigênio) é necessário à respiração aeróbica, sendo importante para suprir o ATP demandado pela nitrogenase e por outras atividades metabólicas tanto na bactéria quanto nas células vegetais.
A regulação do O2, é realizada, em grande parte, pela presença de uma proteína heme que se liga ao oxigênio, a leg-hemoglobina. Esta proteína é encontrada em concentrações elevadas no citossol (citoplasma) das células infectadas e confere uma cor rosada à região central do nódulo, sendo produzida parcialmente pelo bacterióide (porção heme) e e parcialmente pela planta (porção globina).
Acredita-se que a a leg-hemoglobina tampona a concentração de O2 dentro do nódulo, permitindo a a respiração sem que ocorra a inibição da enzima nitrogenase. A leg-hemoglobina também atua como transportadora de O2; facilitando a difusão deste gás para os bacterióides.
Etapas
Amonificação ou Amonização
A primeira etapa do ciclo do nitrogênio, a fixação biológica, ocorre através de um processo enzimático, onde o nitrogênio atmosférico (N2) é reduzido à amônia (NH3), por intermédio da ação de microrganismos de vida livre, simbióticos ou associados aos vegetais, como mostra a equação química abaixo:
N2 + 8e– +8H++16ATP → Nitrogenase → 2NH3 +H2 +16ADP+16Pi
De acordo com Vieira (2017), a fixação do nitrogênio atmosférico está relacionado à presença da ligação covalente tripla entre as moléculas, o que torna este gás altamente estável em temperatura ambiente.
O rompimento desta tripla ligação pelos microrganismos (quimiosintetizantes nitrificantes) do solo requer a presença da enzima nitrogenase (sintetizada pelas bactérias, cujo gene esta num plasmidio em Rhizobium e no próprio cromossomo bacteriano em Bradyrhizobium), além de ATP. Essa enzima requer minerais como molibdênio, ferro e sulfatos para seu funcionamento, pois possui grupos prostéticos de molibdênio, ferro e sulfato em sua estrutura, motivo pelo qual esses organismos devem ser fronecidos ao soo caso sua concentração seja baixa.
Os microrganismos fixadores de N2 podem existir como organismos de vida livre e em associações com diferentes graus de complexidade com as plantas. Estes microrganismos podem ser divididos em:
1. Fixadores não-simbióticos ou de vida livre;
2. Fixadores associativos, que formam uma relação casual e pobremente estruturada com raízes ou porções aéreas das plantas; e
3. Fixadores simbióticos que fixam o N2 em associações organizadas com plantas superiores (RAVEN, EVERT & EICHORN, 2007, VIEIRA, 2017).
Dentre os fixadores simbióticos, existem alguns gêneros de bactérias que se destacam por sua eficiência e especificidade com plantas leguminosas, os chamados rizóbios, tais como:
Rhizobium,
Sinorhizobium (Ensifer),
Mesorhizobium,
Azorhizobium e
Bradyrhizobium.
Para que a simbiose ocorra, alguns fatores são determinantes, como disponibilidade de nutrientes (fósforo, cobalto, molibdênio, níquel), pH do solo (se baixo, pode inibir a ação das bactérias), temperatura e regime hídrico adequado.
Nitrificação
A nitrificação consiste na oxidação do amônio (NH4+) a nitrato (NO3–), onde as bactérias nitrificantes utilizam a amônia como fonte de energia e o dióxido de carbono (CO2) como fonte de carbono (NANES, 2017). Este processo ocorre apenas na presença de oxigênio, por ser realizado por bactérias aeróbias. Esta etapa se divide em duas partes, como demonstram as equações químicas a seguir:
Nitrosação
2NH3 + 3O2 → 2H+ + 2NO2– + 2H2O + energia (eq. 1)
Nitratação ou Nitração
2NO2– + O2 → 2NO3– + energia (eq. 1)
A equação “1” corresponde ao processo de nitrosação, que ocorre através da ação de bactérias do gênero Nitrosomonas. Neste momento transcorre a formação do nitrito, que será oxidado a nitrato na equação “2”, chamada de nitratação, realizada por espécies do gênero Nitrobacter. De acordo com Vieira (2017), as bactérias que participam da nitrificação utilizam compostos nitrogenados como fonte de energia e CO2 como fonte de carbono, que necessitam de O2 nos seus processos metabólicos e possuem crescimento muito lento.
Dentre os fatores que afetam positiva ou negativamente o processo de nitrificação, temos a aeração do solo, visto que as bactérias responsáveis por esta etapa são aeróbias; temperatura (ideal entre 26 ºC e 32 ºC); umidade do solo; calagem e a utilização de fertilizantes.
As relações C/N também influenciam diretamente o processo de nitrificação. Segundo Victoria et al. (1992), quando elevadas, causam imobilização de N mineral, pelo menos temporariamente, cessando a nitrificação por falta de substrato e podendo causar deficiência de nitrogênio aos vegetais. De acordo com Moreira e Siqueira (2006), em um solo que oferece condições favoráveis à nitrificação, o nível de nitrato encontra-se razoavelmente alto, enquanto a relação C/N do solo, baixa. Ainda de acordo com estes autores, quando se adiciona quantidade elevada de resíduos orgânicos com alta relação C/N, os microrganismos que atuam na decomposição da matéria orgânica se tornam altamente ativos, produzindo grandes quantidades de CO2, o que faz com que o nitrato e o amônio praticamente desapareçam do solo.
A desnitrificação é o fenômeno de transformação de nitratos e outras substâncias em gás nitrogênio (N2) pela ação de bactérias desnitrificantes. No solo, além das bactérias de nitrificação existem outros tipos, como as Pseudomonas denitrificans. Na ausência de oxigênio atmosférico, essas bactérias usam o nitrato para oxidar compostos orgânicos (respiração anaeróbia). Através da desnitrificação, uma parte dos nitratos do solo é volta novamente à atmosfera na forma de gasosa, concluindo o ciclo e equilibrando a taxa de nitrato no solo.
A desnitrificação pode ser representada pela reação:
5C6H12O6 + 24NO-3 + 24H+ → 30CO2 + 42H2O + 12N2 + Energia
(glicose) (gás nitrogênio)
A fixação do nitrogênio realizada pelas bactérias, algas azuis e fungos que vivem livres no solo ou associados às raízes de plantas é denominada de fixação biológica ou biofixação. As bactérias do gênero Rhizobium, algas azuis do gênero Anabaena e Nostoc e certos fungos são organismos fixadores de nitrogênio que vivem associados a plantas, principalmente a leguminosas.
A amônia pode ser produzida por dois tipos de biofixadores de vida livre: bactérias dos gêneros Azotobacter (aeróbias) e Clostridium (anaeróbias).
Quando qualquer ser vivos morre os decompositores começam a atuar sobre sua carcaça (atuam sobre a matéria orgânica nitrogenada: aminoácidos livre, proteinas, DNA, RNA, clorofilas etc), liberamdo diversos resíduos para o ambiente, entre eles a amônia (NH3). Combinando-se com a água do solo, a amônia forma hidróxido de amônio que oxidand-se forma o íon amônio (NH4+) e hidroxila. Esse processo é denominado de amonização:
Amonificação ou Amonização
A oxidação dos íons amônio produz nitritos como resíduos nitrogenados, que por sua vez são liberados para o ambiente ou oxidados a nitrato. A conversão dos íons amônio em nitrito e nitrato é conhecida por nitrificação, que ocorre pela ação de bactérias nitrificantes (Nitrosomas, Nitrosococus, Nitrobacter). O processo de nitrificação pode ser dividido em duas etapas:
a) Nitrosação
A amônia é transformada em nitrito (NO2–):
b) Nitração ou nitratação
Ocorre a transformação do íon nitrito em íon nitrato (NO3–):
Os nitratos, quando liberados para o solo, podem ser absorvidos e metabolizados pelas plantas. Assim, o ciclo do nitrogênio envolve três processos:
1) Anomificação ou amonização: quando o N2 é oxidado a amônio
2) Nitrosação: 2NH3 + 3O2 → 2H+ + 2NO2– + 2H2O + energia
conversão de amônia em nitritos (tóxico à planta);
3) Nitração: 2NO2– + O2 → 2NO3– + energia
conversão de nitritos em nitratos (forma assimilável pela planta);
4) Nitrificação: conversão de íons amônio em nitratos.
As bactérias nitrificantes são quimioautróficas, ou seja, utilizam-se da energia liberada na nitrificação para sintetizar as suas substâncias orgânicas. Por meio de excreção ou da morte, os produtos nitrogenados dos organismos são devolvidos ao ambiente. Os excretas nitrogenados eliminados para o ambiente, como uréia e ácido úrico, são transformados em amônia pela ação de bactérias e fungos decompositores.
Outros compostos nitrogenados, como proteínas, por exemplo, são degradados por ação de bactérias e fungos, transformando-os em amônia. A decomposição que se apresenta como produto final é denominada amonificação.
A amônia produzida pelos fixadores ou pela amonificação pode ser aproveitada pelas bactérias nitrificantes ou ser transformada em N2 livre, desprendendo-se para a atmosfera. Essa devolução de nitrogênio para a atmosfera é conhecida por desnitrificação e é comumente realizada pelas bactérias desnitrificantes (Pseudomonas denitificans). Aparentemente indesejável, a desnitrificação é necessária porque, se não ocorresse, a concentração de nitratos no solo aumentaria de maneira desastrosa.
O ciclo do nitrogênio, assim como o do carbono, é um ciclo gasoso. Apesar dessa similaridade, existem algumas diferenças notáveis entre os dois ciclos:
1) a atmosfera é rica em nitrogênio (78%) e pobre em Carbono (0,032%);
2) apesar dessa abundância, somente um dois grupos de organismos conseguem utilizar o nitrogênio gasoso (Eubacteria e os Fungi);
3) o envolvimento biológico no ciclo do nitrogênio é muito mais extenso do que no ciclo do carbono.
RESUMO
Fixação de nitrogênio
A fixação de nitrogênio é o processo pelo qual o nitrogênio gasoso (N2) é convertido em amônia (NH3 ou NH4 +) por meio de fixação biológica ou nitrato (NO3-) por meio de processos físicos de alta energia. N2 é extremamente estável e uma grande quantidade de energia é necessária para quebrar as ligações que unem os dois átomos de N. O N2 pode ser convertido diretamente em NO3- por meio de processos que exercem uma enorme quantidade de calor, pressão e energia. Esses processos incluem combustão, ação vulcânica, descargas atmosféricas e meios industriais.
No entanto, uma maior quantidade de nitrogênio biologicamente disponível é gerada naturalmente por meio da conversão biológica de N2 em NH3 / NH4 +. Um pequeno grupo de bactérias e cianobactérias são capazes, usando a enzima nitrogenase, de quebrar as ligações entre o nitrogênio molecular e combiná-lo com o hidrogênio.
A nitrogenase só funciona na ausência de oxigênio. A exclusão do oxigênio é realizada de várias maneiras. Algumas bactérias vivem sob camadas de limo que exclui o oxigênio nas raízes de certas plantas.
Uma das bactérias mais importante do solo, o rizóbio, vive em zonas livres de oxigênio em nódulos nas raízes de leguminosas e algumas outras plantas lenhosas. As cianobactérias filamentosas aquáticas utilizam células especiais que podem excluir o oxigênio, que se chamam heterocistos.
Nitrificação
A nitrificação é um processo de duas etapas em que NH3 / NH4+ é convertido em NO3-.
Primeiro, as bactérias do solo Nitrosomonas e Nitrococcus convertem NH3 em NO2-, e então outra bactéria do solo, Nitrobacter, oxida NO2- em NO3-.
Essas bactérias ganham energia por meio dessas conversões, que requerem oxigênio para ocorrer, e usam o CO2 como fonte de carbono.
A nitrificação é um processo químico-biológico de formação de nitrito no solo pela ação conjunta de bactérias quimiossintetizantes nitrificantes, pela ação de conversão da amônia em nitrato, ocorrendo em duas etapas.
Nitrosação
A maior parte da amônia não é absorvida pelas plantas, sendo oxidadas em nitrito pelas bactérias nitrosas, que pertencem aos gêneros: Nitrossomonas, Nitrosococus e Nitrosolobus, utilizando a energia liberada nessa oxidação para produzir compostos orgânicos.
A reação pode ser expressa da seguinte forma:
2NH3 + 3O2 → 2H+ + 2NO-2 + 2H2O + Energia
(amônia) (nitrito)
Nitratação
Os nitratos formados pelas bactérias nitrosas são liberados no solo e oxidados por outras bactérias quimiossintéticas chamadas nítricas (do gênero Nitrobacter).
Nessa reação formam-se os nitratos, absorvidos e utilizados pelas plantas na fabricação de suas proteínas e de seus ácidos nucleicos.
2NO-2 + O2 → 2NO-3 + Energia
(nitrito) (nitrato)
Esses elementos químicos são repassados aos demais organismos por meio das relações ecológicas mantidas através da cadeia alimentar, de acordo com os níveis tróficos (produtor, consumidor primário, consumidor secundário, terciário...).
A desnitrificação é o fenômeno de transformação de nitratos e outras substâncias em gás nitrogênio (N2) pela ação de bactérias desnitrificantes. No solo, além das bactérias de nitrificação existem outros tipos, como as Pseudomonas denitrificans. Na ausência de oxigênio atmosférico, essas bactérias usam o nitrato para oxidar compostos orgânicos (respiração anaeróbia). Através da desnitrificação, uma parte dos nitratos do solo é volta novamente à atmosfera na forma de gasosa, concluindo o ciclo e equilibrando a taxa de nitrato no solo.
A desnitrificação pode ser representada pela reação:
5C6H12O6 + 24NO-3 + 24H+ → 30CO2 + 42H2O + 12N2 + Energia
(glicose) (gás nitrogênio)
Assimilação
A assimilação é o processo pelo qual as plantas e animais incorporam o NO3- e a amônia formados através da fixação de nitrogênio e nitrificação. As plantas absorvem essas formas de nitrogênio por meio de suas raízes e as incorporam às proteínas vegetais, à clorofila e aos ácidos nucléicos. Os animais, herbívoros são então capazes de utilizar o nitrogênio dos tecidos vegetais para sua nutrição.
Ammonificação
A assimilação produz grandes quantidades de nitrogênio orgânico, incluindo proteínas, aminoácidos e ácidos nucléicos.
A amonificação é a conversão de nitrogênio orgânico em amônia.
A amônia é produzida quando uma carcaça se decompõem pela ação de bactérias saprófitas voltando para o meio ambiente e então fica disponível para nitrificação ou assimilação.
Desnitrificação
A desnitrificação é a redução de NO3- a N2 gasoso por bactérias anaeróbias. Esse processo ocorre apenas onde há pouco ou nenhum oxigênio, como no fundo do solo próximo ao lençol freático, em brejos e pântanos.
Outras bactérias fixadoras de N
Azotobacter spp, Azotobacter chroococcum, A. vinelandii.
A Azotobacter é um género de bactérias esféricas (cocus) ou ovais Gram negativas geralmente móveis, são relativamente grandes 1–2 μm de diâmetro, que formam esporos de paredes espessas que podem sobreviver em solos secos por mais de 24 anos. As células de Azotobacter sp podem produzir grandes quantidades de mucosidade em sua cápsula. São aeróbicas, de vida livre, e vivem principalmente nos solos, onde desempenham um importante papel no ciclo do nitrogénio.





























































0 comentários:
Postar um comentário