COVID-19
SARS-CoV & MERS-CoV
O SARS-CoV-2 (ou 2019-nCoV) é o sétimo coronavírus conhecido que infecta humanos. Os vírus SARS-CoV, MERS-CoV e SARS-CoV-2 podem causar doença grave, enquanto HKU1, NL63, OC43 e 229E estão associados a sintomas leves. Nesta seção do blog vamos estudar a origem natural do vírus da COVID-19, a organização do genoma do SARS-CoV-2 a partir da análise comparativa de dados genômicos. Ofereceremos uma perspectiva sobre as características notáveis do genoma SARS-CoV-2 e discutiremos cenários onde essses notáeis vírus poderiam ter surgido. Análises científicas mostram claramente que o SARS-CoV-2 não é uma construção de laboratório ou um vírus propositalmente manipulado para causar doença. Este vírus pertence à linhagem B do gênero β-coronavírus (Zhu et al., 2020) e compartilha características importantes com outros coronaviírus que infectam mamíferos e aves. (ver trabalho de Andersen et al., 2020).
a) Mutações em resíduos da proteína spike (S) do SARS-CoV-2, em contato. A proteína Spike do SARS-CoV-2 (barra vermelha no topo) foi comparada com os coronavírus tipo SARS-CoV mais próximos (mais estreitamente relacionados) e com o próprio SARS-CoV. Os resíduos-chave na proteína S que fazem contato com o receptor ACE2 são marcados com caixas azuis no SARS-CoV-2 e vírus relacionados, incluindo SARS-CoV (cepa Urbani). b) Aquisição do sitio de clivagem polibásico e glicanos O-ligados. Ambos, os sitios polibásicos de clivagem e os três glicanos-O-ligados preditos são exclusivos do SARS-CoV-2, e não foram visto previamente em betacoronavírus da linhagem B. As sequências mostradas são do NCBI GenBank, códigos de acesso MN908947, MN996532, AY278741, KY417146 e MK211376. As sequências do coronavírus de pangolin são um consenso gerado a partir de SRR10168377 e SRR10168378 (NCBI BioProject PRJNA573298) 29,30.
a) Mutations in contact residues of the SARS-CoV-2 spike protein. The spike protein of SARS-CoV-2 (red bar at top) was aligned against the most closely related SARS-CoV-like coronaviruses and SARS-CoV itself. Key residues in the spike protein that make contact to the ACE2 receptor are marked with blue boxes in both SARS-CoV-2 and related viruses, including SARS-CoV (Urbani strain). b) Acquisition of polybasic cleavage site and O-linked glycans. Both the polybasic cleavage site and the three adjacent predicted O-linked glycans are unique to SARS-CoV-2 and were not previously seen in lineage B betacoronaviruses. Sequences shown are from NCBI GenBank, accession codes MN908947, MN996532, AY278741, KY417146 and MK211376. The pangolin coronavirus sequences are a consensus generated from SRR10168377 and SRR10168378 (NCBI BioProject PRJNA573298)29,30.Acima é mostrada uma árvore de probabilidade máxima IQ-TREE de representantes de vírus únicos de treze espécies e cinco representantes da espécie Coronavírus relacionado à síndrome respiratória aguda grave do gênero Betacoronavirus. A árvore está enraizada com HCoV-NL63 e HCoV-229E, representando duas espécies do gênero Alphacoronavirus. O texto em roxo destaca os vírus zoonóticos com patogenicidade variada em humanos; o texto laranja destaca os vírus respiratórios comuns que circulam em humanos. Os asteriscos indicam duas espécies de coronavírus cujas demarcações e nomes aguardam aprovação do ICTV e, portanto, esses nomes não estão em itálico.
Inicialmente, a classificação dos coronavírus foi amplamente baseada em reatividades sorológicas (cruzadas) para a proteína spike viral, mas agora é baseada em análises de sequências comparativas de proteínas replicativas. A escolha das proteínas e os métodos utilizados para analisá-las evoluíram gradativamente desde o início deste século. O CSG (Coronavirus Study Group) analisa atualmente 3CLpro, NiRAN, RdRp, ZBD e HEL1, dois domínios a menos do que anteriormente usado nas análises conduzidas entre 2009 e 2015. De acordo com nosso conhecimento atual, esses cinco domínios essenciais são os únicos conservados em todos os vírus da ordem Nidovirales. Eles são, portanto, usados para a classificação por todos os grupos de estudo de nidovírus do ICTV (coordenados pelo NSG). (nature, 2021, ncbi, 2020)
As comparações de alfa e betacoronavírus, existentes na literatura identificam duas características genômicas notáveis do SARS-CoV-2:
(i) com base em estudos estruturais e experimentos bioquímicos, o SARS-CoV-2 parece ser otimizado para ligação ao receptor humano ACE2.
(ii) a proteína spike do SARS-CoV-2 tem um site (local) plibásico funcional de clivagem (furina) no limite S1-S2 através da inserção de 12 nucleotídideos, o que adicionalmente leva à aquisição prevista de 3 O-glicanos ligados ao redor do site.
((ii) the spike protein of SARS-CoV-2 has a functional polybasic (furin) cleavage site at the S1–S2 boundary through the insertion of 12 nucleotides8, which additionally led to the predicted acquisition of three O-linked glycans around the site).
Alguns coronavírus podem causar síndromes respiratórias graves, como a síndrome respiratória aguda grave que ficou conhecida pela sigla SARS da síndrome em inglês “Severe Acute Respiratory Syndrome”.
A SARS é causada pelo coronavírus associado à SARS (SARS-CoV), sendo os primeiros relatos na China em 2002. O SARS-CoV se disseminou rapidamente para mais de doze países, na América do Norte, América do Sul, Europa e Ásia, infectando mais de 8.000 pessoas e causando entorno de 800 mortes, antes da epidemia global de SARS ser controlada em 2003. Desde então (2004 até hoje), nenhum caso de SARS tem sido relatado mundialmente.
Em 2012, foi isolado outro novo coronavírus, distinto daquele que causou a SARS no começo da década passada. Esse novo coronavírus era desconhecido como agente de doença humana até sua identificação, inicialmente na Arábia Saudita e, posteriormente, em outros países do Oriente Médio, na Europa e na África. Todos os casos identificados fora da Península Arábica tinham histórico de viagem ou contato recente com viajantes procedentes de países do Oriente Médio: Arábia Saudita, Catar, Emirados Árabes e Jordânia.
Pela localização dos casos, a doença passou a ser designada como síndrome respiratória do Oriente Médio, cuja sigla é MERS, do inglês “Middle East Respiratory Syndrome” e o novo vírus nomeado coronavírus associado à MERS (MERS-CoV).
HISTÓRIA
Os coronavírus tem uma grande diversidade de hospedeiros entre aves e mamíferos. Esses vírus estão associados com doenças respiratórias, entéricas, hepáticas e neurológicas. É um vírus envelopado.
Genoma: possui o maior genoma entre os vírus de RNA (~29 pares de bases) de fita simples de sentido positivo ssRNA(+). Esse vírus usa uma enzima de replicase de DNA (ou uma polimerase de RNA dependente de RNA) em sua replicação (não apresentando uma transcriptase reversa).
Foi identificado pela primeira vez na década de 60, infectando humanos (cepa HCoV-229E), e sabe-se que pode infectar um grande número de animais, principalmente mamíferos e aves (mas não são limitados a esses, há registro de que repteis também sejam infectados por coronavírus).
Ordem: Nidovirales
Família: Coronaviridae
Subfamília: Orthocoronavirinae
Gêneros:
Alphacoronavirus, Betacoronavirus: predomina em mamíferos
Gammacoronavirus, Deltacoronavirus: predominante em aves.
Estrutura do vírion SARS-CoV-2
Partícula de β-coronavírus e seu genoma (A) A partícula (vírion) de β-coronavírus. O β-coronavírus é um vírus cujo genoma é de RNA de fita simples de sentido positivo, não segmentado e com envelope, em um tamanho que varia de 29,9 kb. O vírion possui um nucleocapsídio composto de RNA genômico e proteína nucleocapsídio fosforilado (N), que esta ancorada dentro da bicamada fosfolipídicas e coberta pelas glicoproteína "spike" trímeras (S). A proteína de membrana (M), hemaglutinina esterase (HE) e a proteína do envelope (E) estão localizadas entre as proteínas S no envelope do vírus. (B) sequências terminais 5' e 3' do genoma SARS-CoV-2. A ordem dos gene da é 5' replicase ORF1ab-S-envelope (E) membrana (M) N-3'. ORF3ab, ORF6, ORF7ab, ORF8, ORF9ab e ORF10 estão localizados nas posições mostradas na figura acima.
β-coronavirus particle and genome [9] (A) The β-coronavirus particle. β-coronavirus is an enveloped, nonsegmented, positive-sense single-stranded RNA virus genome in a size ranging from 29.9 kb. The virion has a nucleocapsid composed of genomic RNA and phosphorylated nucleocapsid (N) protein, which is buried inside phospholipid bilayers and covered by the spike glycoprotein trimmer (S). The membrane (M) protein hemagglutinin esterase (HE) and the envelope (E) protein are located among the S proteins in the virus envelope. (B) 5 ′ and 3 ′ terminal sequences of the SARS-CoV-2 genome. The order gene is 5′-replicase ORF1ab-S-envelope (E)-membrane (M)-N-3′. ORF3ab, ORF6, ORF7ab, ORF8, ORF9ab, and ORF10 are located at the predicted positions shown in the picture.
REPLICAÇÃO DO NOVO CORONAVIRUS:
SARS-CoV-2
O vírion e o genoma do SARS-Cov-2
Os coronavírus são vírus de RNA de fita simples com envelope relativamente grande, sentido positivo (aproximadamente 29,9 kb) ssRNA(+). O sentido positivo possibilita que esse RNA ao entrar no citoplasma da célula hospedeira, seja imediatamente transcrito em suas proteínas estruturais e não estruturais.
O genoma SARS-CoV-2 codifica quatro proteínas estruturais e outras proteínas acessórias ou não estruturais (nsp), incluindo uma replicase pp1a-pp1ab viral, a protease semelhante a 3C (3CLpro), a protease semelhante à papaína (PLpro) e a RNApolimerase dependente de RNA, ou RdRp) (Fehr & Perlman, 2015, Zhou, P. et al., 2020).
As proteínas estruturais do SARS-CoV-2 são listadas a seguir.
1) Proteína de superfície Spike (S) forma grandes estruturas triméricas que são essenciais para a entrada nas células hospedeiras após a ligação ao receptor e fusão da membrana. As proteínas spike são direcionadas por anticorpos neutralizantes do hospedeiro.
2) O Proteína do envelope (E) está presente apenas em pequenas quantidades e provavelmente forma canais iônicos. As proteínas E não são necessariamente usadas para a replicação viral, mas são essenciais para a infectividade e patogênese.
3) Proteína Matriz/Membrana (M) é a proteína estrutural mais abundante do vírus. As proteínas M são responsáveis pela curvatura da membrana do envelope viral, notadamente por meio de sua interação com as proteínas E.
4) O Proteínas do nucleocapsídeo (N) se liga ao genoma do RNA viral e garante a manutenção do RNA em uma conformação de "contas em um fio", ou um colar de pérolas. Observe a figura a seguir que detalha essa conformação do vírus.
O Coronavírus SARS-CoV-2 é um vírus de RNA de cadeia simples, com cerca de 29.903 nucleotídeos, aproximadamente 29,9 kB, na qual se reconhece 13 genes que codificam para 14 proteínas conhecidas.
Apresenta uma longa ORF1ab (ORF: Open Reading Frame) na posição 5’ que codifica proteínas ORF não estruturais, seguido de quatro genes para proteínas estruturais: proteína de superfície (S), proteína do envelope (E), proteína da membrana (M) e proteína do nucleocapsídeo (N).
Estrutura do vírion SARS-CoV-2
Os coronavírus patogênicos humanos (coronavírus da síndrome respiratória aguda grave (SARS-CoV e SARS-CoV-2) se ligam às células-alvo através da enzima conversora de angiotensina 2 (ACE2), que é expressa pelas células epiteliais do pulmão, intestino, rim, e vasos sanguíneos.
A expressão da ECA2 é substancialmente aumentada em pacientes com diabetes tipo 1 ou tipo 2, que são tratados com inibidores da ECA e bloqueadores dos receptores da angiotensina II tipo I (BRA).
A hipertensão também é tratada com inibidores da ECA e BRA, o que resulta em uma regulação positiva da ACE2. A ACE2 também pode ser aumentada por tiazolidinedionas e ibuprofeno. Esses dados sugerem que a expressão da ACE2 é aumentada no diabetes e o tratamento com inibidores da ECA e BRA aumenta a expressão da ACE2.
Consequentemente, o aumento da expressão de ACE2 facilitaria a infecção por COVID-19. Portanto, supomos que o tratamento do diabetes e da hipertensão com medicamentos estimulantes da ECA2 aumente o risco de desenvolver COVID-19 grave e fatal.
Os genomas de vírus de RNA de fita positiva, como o coronavírus, pode atuar como mRNA (RNA mensageiro) e ser diretamente traduzido em proteínas no citoplasma das células hospedeiras. Os intermediários de RNA de fita negativa também são produzidos por coronavírus que servem como moldes para: síntese de fita positiva de RNA genômico, que é então empacotado pelas proteínas estruturais para montar a prole do vírion; e transcritos de RNA subgenômicos.
Vários Open Reading Frame “sequencia/quadros de leitura aberta” (ORFs) foram distinguidos dentro da sequência do "corpo" do genoma SARS-CoV-2, correspondendo a elementos estruturais virais (proteínas N, S, E e M) e genes acessórios.
A proteína N, ou nucleocapsídeo, encapsula o genoma, enquanto as proteínas S (spike), E (envelope) e M (membrana) constituem o envelope de bicamada lipídica circundante. O envelope está possivelmente reduzido a canais iônicos.
A proteína S é importante pois permite a infecção viral através do reconhecimento do receptor ACE-2 do hospedeiro e fusão do envelope viral com a membrana celular.
A ORF mais 5', ORF1a/1b, também conhecida como o gene replicase/transcriptase é relatado em coronavírus para codificar polimerases para a síntese de RNA viral e outras proteínas não estruturais (nsps) (por exemplo, para cauda poli-A).
Isso significa dizer que o Sars-CoV-2, compartilha com outros 29 tipos de vírus algumas características como:
(i) RNA de fita simples positiva, (+) ssRNA(+),
(ii) São os mais abundantes do planeta, com um genoma linear que varia entre 2,3 e 32 kpb (pares de bases),
(iii) O genoma viral poder ser constituído por um único segmento ou por várias moléculas de RNA.
(iv) São vírus com capsídeos de simetria icosaédrica ou helicoidal.
(v) Os vírions deste grupo podem ser envelopados ou não (vírus nus).
(vi) No envelope apresentam peplômeros ou espículas (que em imagens de microscopia eletrônica assemelham-se a coroa solar).
(vii) A extremidade 5' do genoma possue um cap 5' e a 3' uma cauda poli-A.
(viii) Seu envelope (adquirido ao brotar da membrana do retículo endoplasmático e do complexo de Golgi), invariavelmente apresenta glicoproteínas específicas do vírus, chamadas de S e M.
(ix) A glicoproteína S constitui as grandes projeções superficiais do envelope (que forma a coroa nas microfotografias eletrônicas).
(x) A glicoproteína transmembranar M é uma proteína que atravessa três vezes a membrana do envelope;
(xi) O nucleocapsideio apresenta uma fosfoproteína, N.
(xii) Alguns vírus desse grupo possuem peplômeros HE com atividade de hemaglutinina mais acetilesterase.
Todo o ciclo de replicação ocorre no citoplasma, maturação de proteínas no reticulo endoplasmático e no complexo de Golgi e é relativamente lento.
Após a adsorção, a penetração (endocitose) e o desnudamento, a molécula de RNA do vírion que entra é traduzida diretamente, um dos produtos sendo uma polimerase de RNA, RNA dependente (RdRp) que, em seguida, transcreve um RNA completo de sentido negativo, do qual é transcrito um "conjunto aninhado" 3'-co-terminal de mRNAs subgenômicos.
Esse conjunto aninhado compreende de cinco a sete espécies sobrepostas de mRNAs que se estendem por diferentes comprimentos a partir de um terminal 3' comum.
O RNA genômico e todos os mRNAs têm uma sequência líder de 5' idêntica de cerca de 72 nucleotídeos.
Consequentemente, o conjunto aninhado de mRNAs é formado pelo mecanismo incomum de unir dois RNAs não contíguos. A união da sequência líder 5' com a parte restante de cada mRNA ocorre durante a transcrição.
Resumindo: após a liberação do RNA genômico de fita (+) no citoplasma, é sintetizada uma RNA polimerase dependente de RNA, que transcreve um RNA de fita (-) completo, a partir do qual são sintetizados (a) novo RNA genômico, (b) uma série sobreposta de mRNAs subgenômicos e (c) o RNA líder.
Transcrição e tradução do mRNA do SARS-CoV-2
Modified from K. V. Holmes, In "Fields Virology" (B. N. Fields et al., eds.), 2nd Ed., p. 847. Raven, New York, 1990.1
O RNA genômico e os mRNAs são limitados e poliadenilados (linha em ziguezague na figura acima) e formam um "conjunto aninhado" com extremidades 3 'comuns e uma sequência líder comum na extremidade 5'.
Apenas a sequência única dos mRNAs na extremidade 5' é traduzida, para produzir várias proteínas não estruturais (NS ou nsp) e quatro proteínas estruturais: M (EI), glicoproteína transmembranar; S (E2), glicoproteína peplomérica; N, nucleoproteína; e em alguns coronavírus HE (E3), glicoproteína hemaglutinina-esterase.
A maturação e a montagem do vírion ocorrem no retículo endoplasmático rugoso e no Golgi, e os vírions são liberados por exocitose.
Estudos recentes indicam que a patogenicidade e a virulência do SARS-CoV-2, está associada a alta afinidade com os receptores de enzima conversora da angiotensina 2 (ACE2), uma aminopeptidase ligada à membrana que é altamente expressa no coração e nos pulmões, mas encontrada na superfície de vários tipos de células humanas, sendo que esta ligação, eficiente e estável, parece também facilitar a dispersão viral de pessoa-para-pessoa.
A ACE2 é uma proteína transmembrana expressa na superfície de diversas células do corpo, como o epitélio do sistema respiratório.
Vários estudos já demonstraram a relação entre a proteína ACE2 com os mecanismos de entrada de alguns coronavírus, como o HCoV-NL63, o SARS-CoV e o novo SARS-CoV-2 (causador da COVID-19). (Damasio, 2020).
Qual a função do gene ACE2 em nosso organismo?
A princípio, o gene da ACE2 é responsável pela expressão da proteína de mesmo nome, ACE2 (da sigla em inglês: Angiotensin-Converting Enzyme 2).
Essa proteína está expressa na superfície das células, ela nada mais é, do que um homólogo da já conhecida ACE (em português: ECA), responsável pela regulação da pressão arterial dentro do Sistema Renina-Angiotensina.(Damasio, 2020).
A proteína variante ACE2, descoberta no ano 2000, é muito semelhante em estrutura (cerca de 42%) mas faz o papel inverso da ACE.
Enquanto a ACE faz vasoconstrição e consequentemente o aumento da pressão arterial, a ACE2 promove a vasodilatação e diminui a pressão arterial. Dessa forma é feita a regulação do Sistema Renina-Angiotensina.
Apesar de a ACE2 ser a porta de entrada do vírus nas células, foi mostrado em alguns artigos que a ação da ACE2 tem efeito protetor contra danos pulmonares causados por infecções virais.
Consequentemente, os estudos mostraram que a diminuição nos níveis dessa proteína pode causar ainda mais danos aos pulmões de quem está infectado. (Damasio, 2020).
Coronavirus polyprotein processing and non structural proteins
O processamento da poliproteína de coronavírus e os domínios de proteínas não estruturais (nsp) são ilustrados para coronavírus relacionados com a síndrome respiratória aguda grave.
A clivagem proteolítica das poliproteínas pp1a e pp1ab é facilitada por proteases virais que residem em nsp3 (PLpro) e nsp5 (Mpro).
PLpro libera proteoliticamente nsp1, nsp2, nsp3 e o terminal amino de nsp4 das poliproteínas pp1a e pp1ab (indicadas pelas setas azuis).
Mpro libera proteoliticamente o nsp5-16 e o terminal carboxi do nsp4 das poliproteínas pp1a e pp1ab (indicadas pelas setas vermelhas).
Domínios conservados e funções conhecidas são esquematicamente representados para nsp1–16. DMV, vesícula de membrana dupla; DPUP, Domínio que precede Ubl2 e PLpro; EndoU, endoribonuclease; ExoN, exoribonuclease; HEL, helicase; Mac I-III, macrodomínios 1–3; Mpro, protease principal; NiRAN, nucleotidiltransferase associada a nidovírus RdRP; NMT, guanosina N7-metiltransferase; OMT, ribose 2'-O-metiltransferase; PLpro, protease semelhante à papaína; Pr, primase ou 3' terminal Adenilil-transferase, RdRP, RNA polimerase dependente de RNA; TM, domínios transmembranares; Ubl, domínio do tipo ubiquitina; Domínio; Y, dominio Y e CoV-Y; ZBD, domínio da ligação de zinco. (V'kovski et al. 2020)
Coronavirus polyprotein processing and domains of non-structural proteins (nsp) are illustrated for severe acute respiratory syndrome-related coronaviruses. Proteolytic cleavage of the polyproteins pp1a and pp1ab is facilitated by viral proteases residing in nsp3 (PLpro) and nsp5 (Mpro). PLpro proteolytically releases nsp1, nsp2, nsp3 and the amino terminus of nsp4 from the polyproteins pp1a and pp1ab (indicated by the blue arrows). Mpro proteolytically releases nsp5–16 and the carboxy terminus of nsp4 from the polyproteins pp1a and pp1ab (indicated by the red arrows). Conserved domains and known functions are schematically depicted for nsp1–16. DMV, double-membrane vesicle; DPUP, Domain Preceding Ubl2 and PLpro; EndoU, endoribonuclease; ExoN, exoribonuclease; HEL, helicase; Mac I–III, macrodomains 1–3; Mpro, main protease; NiRAN, nidovirus RdRP-associated nucleotidyltransferase; NMT, guanosine N7-methyltransferase; OMT, ribose 2′-O-methyltransferase; PLpro, papain-like protease; Pr, primase or 3′-terminal adenylyl-transferase; RdRP, RNA-dependent RNA polymerase; TM, transmembrane domains; Ubl, ubiquitin-like domain; Y, Y and CoV-Y domain; ZBD, zinc-binding domain.
Representação da transcrição descontinua do genoma do vírus SARS-CoV-2.
Representação esquemática da síntese de RNA coronaviral. O RNA genômico de sentido positivo de comprimento total é usado como um modelo para produzir cópias de sentido negativo de comprimento total para a replicação do genoma e RNAs de sentido negativo subgenômico [sgRNA(–)] para produzir os mRNAs subgenômicos (sgmRNA). É ilustrada a síntese de RNA de fita negativa envolvendo uma mudança de molde de uma sequência reguladora de transcrição do corpo (TRS-B) para o TRS líder (TRS-L) para produzir um sg mRNA. Este processo pode ocorrer em qualquer TRS-B e resultará coletivamente na produção do característico conjunto aninhado de mRNAs coronavirais. (V'kovski et al. 2020)
Schematic depiction of coronaviral RNA synthesis. Full-length positive-sense genomic RNA is used as a template to produce both full-length negative-sense copies for genome replication and subgenomic negative-sense RNAs (–sgRNA) to produce the subgenomic mRNAs (sg mRNA). The negative strand RNA synthesis involving a template switch from a body transcription regulatory sequences (TRS-B) to the leader TRS (TRS-L) is illustrated to produce one sg mRNA. This process can take place at any TRS-B and will collectively result in the production of the characteristic nested set of coronaviral mRNAs.
a) Os genomas de RNA de fita simples (ssRNA) do coronavírus da síndrome respiratória aguda grave (SARS-CoV) e do coronavírus da síndrome respiratória do Oriente Médio (MERS-CoV) codificam duas grandes poliproteínas, pp1a e pp1ab, que são proteoliticamente clivadas em 16 proteínas estruturais (nsps), incluindo protease do tipo papaína (PLpro), protease do tipo 3C (3CLpro), RNA polimerase dependente de RNA (RdRp), helicase (Hel) e exonuclease (ExoN). Outros 9-12 ORFs são codificados através da transcrição de um conjunto aninhado de RNAs subgenômicos. SARS-CoV e MERS-CoV formam partículas esféricas que consistem em quatro proteínas estruturais. O pico da glicoproteína do envelope (S) forma uma camada de glicoproteínas que se projetam do envelope. Duas glicoproteínas transmembranares adicionais são incorporadas no vírion: envelope (E) e membrana (M). Dentro do envelope viral reside o nucleocapsídeo helicoidal, que consiste no genoma do RNA de sentido positivo viral (+RNA) encapsidado pela proteína nucleocapsídeo (N).
Diagrama esquemático que mostra o ciclo de replicação do coronavírus e os estágios nos quais o estresse do ER pode ser induzido durante a infecção pelo coronavírus. A infecção começa com a ligação ao receptor e entrada por fusão da membrana. Após a remoção do revestimento, o RNA genômico é usado como molde para sintetizar genomas de progênie e um conjunto aninhado de RNAs subgenômicos. Os centros de transcrição de replicação estão intimamente associados aos DMVs, que são propostos para serem adotados a partir do ER modificado, possivelmente pelas atividades combinadas das proteínas não estruturais nsp3, nsp4 e nsp6. As proteínas S, E e M são sintetizadas e ancoradas no ER, enquanto a proteína N é traduzida no citosol. A montagem ocorre no ERGIC e os vírions maduros são liberados por meio de vesículas de parede lisa por exocitose. Os três estágios que presumivelmente induzem estresse ER são destacados com estrelas numeradas, a saber: (1) formação de DMVs, (2) produção massiva e modificação de proteínas estruturais, e (3) esgotamento da membrana ER durante o brotamento.
Schematic diagram showing the replication cycle of coronavirus and the stages in which ER stress may be induced during coronavirus infection. Infection starts with receptor binding and entry by membrane fusion. After uncoating, the genomic RNA is used as a template to synthesize progeny genomes and a nested set of subgenomic RNAs. The replication transcription centers are closely associated with DMVs, which are proposed to be adopted from the modified ER, possibly by the combined activities of non-structural proteins nsp3, nsp4, and nsp6. The S, E, and M proteins are synthesized and anchored on the ER, whereas the N protein is translated in the cytosol. Assembly takes place in the ERGIC and mature virions are released via smooth-walled vesicles by exocytosis. The three stages that presumably induce ER stress are highlighted with numbered star signs, namely: (1) formation of DMVs, (2) massive production and modification of structural proteins, and (3) depletion of ER membrane during budding.
b) Após a entrada do vírus na célula hospedeira, o RNA viral não é revestido no citoplasma. ORF1a e ORF1ab são traduzidos para produzir pp1a e pp1ab, que são clivados pelas proteases e que são codificadas por ORF1a para produzir 16 nsps que formam o complexo RNA replicase-transcriptase. Esse complexo localiza-se nas membranas intracelulares modificadas que são derivadas do retículo endoplasmático rugoso (ER) na região perinuclear e direciona a produção de RNAs de sentido negativo (-RNAs) por meio de replicação e transcrição. (V'kovski et al. 2021)
Durante a replicação, cópias de RNA de comprimento total (-) do genoma são produzidas e usadas como modelos para genomas de RNA de comprimento total (+). Durante a transcrição, um subconjunto de 7 a 9 RNAs subgenômicos, incluindo aqueles que codificam todas as proteínas estruturais, é produzido através da transcrição descontínua. Nesse processo, os RNAs subgenômicos (-) são sintetizados combinando comprimentos variados da extremidade 3' do genoma com a sequência líder 5' necessária para a tradução. Esses RNAs subgenômicos (-) são então transcritos em mRNAs subgenômicos (+).
Embora os diferentes mRNAs subgenômicos possam conter várias ORFs, apenas a primeira ORF (a mais próxima da extremidade 5') é traduzida. As proteínas estruturais resultantes são montadas no envelope e nucleocapsídeo viral no compartimento intermediário ER-Golgi (ERGIC), seguido pela liberação do vírion nascente da célula infectada.
a) The single-stranded RNA (ssRNA) genomes of severe acute respiratory syndrome coronavirus (SARS-CoV) and Middle East respiratory syndrome coronavirus (MERS-CoV) encode two large polyproteins, pp1a and pp1ab, which are proteolytically cleaved into 16 non-structural proteins (nsps), including papain-like protease (PLpro), 3C-like protease (3CLpro), RNA-dependent RNA polymerase (RdRp), helicase (Hel) and exonuclease (ExoN). An additional 9–12 ORFs are encoded through the transcription of a nested set of subgenomic RNAs. SARS-CoV and MERS-CoV form spherical particles that consist of four structural proteins. The envelope glycoprotein spike (S) forms a layer of glycoproteins that protrude from the envelope. Two additional transmembrane glycoproteins are incorporated in the virion: envelope (E) and membrane (M). Inside the viral envelope resides the helical nucleocapsid, which consists of the viral positive-sense RNA ((+)RNA) genome encapsidated by protein nucleocapsid (N). b) Following entry of the virus into the host cell, the viral RNA is uncoated in the cytoplasm. ORF1a and ORF1ab are translated to produce pp1a and pp1ab, which are cleaved by the proteases that are encoded by ORF1a to yield 16 nsps that form the RNA replicase–transcriptase complex. This complex localizes to modified intracellular membranes that are derived from the rough endoplasmic reticulum (ER) in the perinuclear region, and it drives the production of negative-sense RNAs ((−)RNAs) through both replication and transcription. During replication, full-length (−)RNA copies of the genome are produced and used as templates for full-length (+)RNA genomes. During transcription, a subset of 7–9 subgenomic RNAs, including those encoding all structural proteins, is produced through discontinuous transcription. In this process, subgenomic (−)RNAs are synthesized by combining varying lengths of the 3′ end of the genome with the 5′ leader sequence necessary for translation. These subgenomic (−)RNAs are then transcribed into subgenomic (+)mRNAs. Although the different subgenomic mRNAs may contain several ORFs, only the first ORF (that closest to the 5′ end) is translated. The resulting structural proteins are assembled into the nucleocapsid and viral envelope at the ER–Golgi intermediate compartment (ERGIC), followed by release of the nascent virion from the infected cell.
a) O vírion do coronavírus consiste em proteínas estruturais, principalmente a proteína Spike (S), proteina do envelope (E), proteina de membrana (M), do nucleocapsídeo (N) e, para alguns betacoronavírus, hemaglutinina-esterase (não mostrado). O genoma de RNA de fita simples de sentido positivo ssRNA (+) é encapsidado por N, enquanto M e E garantem sua incorporação na partícula viral durante o processo de montagem. Os trímeros S projetam-se do envelope viral derivado do hospedeiro e fornecem especificidade para os receptores de entrada celular.
b) As partículas de coronavírus se ligam a fatores de fixação celular e as interações S específicas com os receptores celulares, como a enzima conversora de angiotensina 2 (ACE2), juntamente com fatores do hospedeiro, como a serina protease da superfície celular TMPRSS2, promovem a absorção viral e a fusão no membrana celular ou endossomal. Após a entrada (endocitose), a liberação (desnudamento) e o desencapsulamento do RNA genômico de entrada é submetido à tradução imediata de duas grandes estruturas de leitura aberta, ORF1a e ORF1b. As poliproteínas pp1a e pp1ab resultantes são processadas ao mesmo tempo que acontece a tradução e após a tradução (The resulting polyproteins pp1a and pp1ab are co-translationally and post-translationally processed into the individual non-structural proteins nsps) nas proteínas não estruturais individuais (nsps) que formam o complexo de replicação e transcrição viral.
Concordante com a expressão de nsps, a biogênese de organelas de replicação viral consistindo em vesículas de membrana dupla perinuclear características (DMVs)(double membrane vesicle), membranas convolutas (CMs) e pequenas esférulas de membrana dupla abertas (DMSs) criam um microambiente protetor para a replicação de RNA genômico viral e transcrição de mRNAs subgenômicos (sg mRNAs) compreendendo o conjunto aninhado característico de mRNAs de coronavírus.
As proteínas estruturais traduzidas se translocam para as membranas do retículo endoplasmático (ER) e transitam através do compartimento intermediário ER-para-Golgi (ERGIC), onde a interação com o RNA genômico N-encapsidado recém-produzido resulta em brotamento no lúmen dos compartimentos vesiculares secretores.
Finalmente, os vírions são secretados da célula infectada por exocitose. As principais etapas inibidas por compostos que estão sendo validados e que representam alvos antivirais atraentes estão destacadas em vermelho. Sequência de cap poliA, 3'; estrutura de cap 5′; dsRNA, RNA de fita dupla; L, sequência líder; RdRP, RNA polimerase dependente de RNA.
Eventos iniciais durante a infecção de HCoV-NL63.
RETROVÍRUS
VÍRUS HIV/AIDS
Os retrovírus são ribovírus são um grupo de vírus de RNA que se replicam para produzir DNA a partir do RNA.
Usando uma enzima denominada transcriptase reversa.
HIV
O DNA produzido é então incorporado ao genoma do hospedeiro. Normalmente, as informações genéticas seguem um fluxo do DNA para o RNA e então para proteínas, ao inverter esse processo, a transcriptase reversa faz uma cópia de um DNA de fita dupla, denominado DNA complementar (cDNA) a partir do genoma de RNA viral de fita dupla. Todos os retrovírus humanos compartilham várias similaridades em relação à estrutura, à organização genômica e ao modo de replicação. Os retrovírus pertencem à família Retroviridae, que infecta aves e mamíferos (provocado cânceres).
Anteriormente os retrovírus eram organizados em três subfamílias.
Oncovirinae
Compreendendo os atuais gêneros alfa, beta, gama, delta e epsilonvirus, oncogênicos, causam sarcomas e leucemias. Ex.: Vírus do Sarcoma de Rous (galinhas), Vírus da Leucemia de Molonei (roedores), Vírus da leucemia humana de células T (HTLV).
Lentivirinae
Doenças degenerativas de progressão lenta. Ex.: Visnavirus (ovelhas); Vírus da imunodeficiência de símios (SIV); Vírus da imunodeficiência humana (HIV).
Spumavirinae
Não há patologia conhecida a eles associada.
Atualmente a família Retroviridae compreende sete gêneros, são eles e suas respectivas espécies:
Alpharetrovírus: Vírus do sarcoma de Rous que tem galinhas como hospedeiro.
Betaretrovírus: Vírus do tumor mamário de camundongos.
Gammaretrovírus: Vírus da leucemia Murina em camundongos.
Deltaretrovírus: Vírus linfotrópico T humano
Epsilonretrovírus: Vírus do sarcoma dérmico de Walleye
Lentivírus: Vírus da imunodeficiência humana (HIV).
Spumavirus: Vírus espumosos dos símios. (todamateria)
Entre os retrovírus que causam doenças em humanos estão o linfotrópico T humanos (HTLV), que possuem afinidade e infectam as células de defesa do organismo, os Linfócitos T e são classificados em quatro grupos, sendo os dois primeiros os mais estudados: HTLV-I e HTLV-II.
O vírus é transmitido por contato direto com fluidos corporais infectados, por relação sexual desprotegida, agulhas infectadas e por transmissão vertical (de mãe para filho) e a grande maioria dos indivíduos infectados são assintomáticos.
Estima-se que apenas 10% dos indivíduos infectados pelo vírus apresentarão algumas doenças associadas como linfoma/leucemia de células T do adulto, linfoma não Hodgkin de células T e síndrome neurodegenerativa atípica, designada paraparesia espástica tropical ou mielopatia associada ao HTLV-I, e o vírus da imunodeficiência humana (HIV), agente infeccioso que causa a Síndrome da imunodeficiência adquirida (AIDS), que atinge o sistema imunológico dos indivíduos infectados, e impede que o organismo consiga combater infecções.
Os retrovírus, assim como outros vírus, possuem a característica de produzir infecções latentes e persistentes, caracterizadas por longos períodos de incubação e crescimento lento das quantidades de vírus nas células infectadas, isso permite que eles permaneçam assintomáticos por um longo período até que a doença associada comece a apresentar os sintomas.
Ciclo de vida do HIV (1)
Ciclo de vida do HIV (2)
HIV é a sigla em inglês do vírus da imunodeficiência humana. Causador da aids, ataca o sistema imunológico, responsável por defender o organismo de doenças. As células mais atingidas são os linfócitos T CD4+. E é alterando o DNA dessa célula que o HIV faz cópias de si mesmo. Depois de se multiplicar, rompe os linfócitos em busca de outros para continuar a infecção.
Ter o HIV não é a mesma coisa que ter aids. Há muitos soropositivos que vivem anos sem apresentar sintomas e sem desenvolver a doença. Mas podem transmitir o vírus a outras pessoas pelas relações sexuais desprotegidas, pelo compartilhamento de seringas contaminadas ou de mãe para filho durante a gravidez e a amamentação, quando não tomam as devidas medidas de prevenção. Por isso, é sempre importante fazer o teste e se proteger em todas as situações.
Biologia
O HIV é um retrovírus, classificado na subfamília dos Lentiviridae. Esses vírus compartilham algumas propriedades comuns: período de incubação prolongado antes do surgimento dos sintomas da doença, infecção das células do sangue e do sistema nervoso e supressão do sistema imune.
Fases da invasão dos Linfócitos T CD4
Adsorção: Vírus chega no linfócito
Ciclo de retrovírus
O ciclo de vida ou replicação de retrovírus é único em comparação com outros organismos. O ciclo de replicação do retrovírus é concluído nas seguintes fases:
1) Infecção: os retrovírus entram em contato com o hospedeiro e infectam as células normais do corpo hospedeiro.Internalização: o genoma de RNA do retrovírus entra no citoplasma da célula hospedeira.
2) Transcrição reversa: no citoplasma da célula hospedeira, ocorre o processo de transcrição reversa. O cDNA é produzido a partir do genoma do RNA viral com a ajuda de uma enzima conhecida como transcriptase reversa, e o processo é conhecido como transcrição reversa.
O cDNA é então feito entrar no núcleo da célula hospedeira através do complexo de poro nuclear.
3) Integração: o cDNA (DNA viral) agora se integra ao genoma da célula hospedeira com a ajuda de uma enzima conhecida como integrase
4) Expressão gênica viral: genes virais são então transcritos para formar mRNA, que mais tarde sofre tradução para produzir proteínas virais.
5) Liberação de partículas virais: as partículas virais são então liberadas da célula hospedeira pela ruptura da membrana celular da célula hospedeira (fase lítica).
6) A infecção se espalha: as partículas virais liberadas das células hospedeiras infectam então as células hospedeiras vizinhas.
Ciclo viral
Visão geral de um ciclo de replicação viral hipotético:
1) Adsorção; 2) Penetração (entrada); 3) Desnudamento; 4) Síntese viral (Transcrição e tradução); 5) Síntese viral: replicação do genoma; 6) Síntese viral: Montagem e maturação; 7) Liberação.(WP)
Fusão: Neste mecanismo, executado apenas por vírus envelopados, o nucleocapsídeo é liberado no interior da célula mediante a fusão entre o envelope viral e a membrana celular. A entrada por fusão pode ocorrer de duas formas: (1) direta, pela fusão do envelope viral com a membrana plasmática, a partir do meio extracelular, ou (2) indireta, sofrendo uma endocitose inicial com posterior fusão já no interior da célula, como citado anteriormente;
Translocação: por meio da ação de uma proteína receptora, o vírion pode atravessar a membrana por meio de translocação, do ambiente extracelular para o citosol. Este mecanismo é raro e pouco entendido (WP).
Ciclo de um vírus
1) Adsorção
É a ligação de uma molécula presente na superfície da partícula viral com os receptores específicos da membrana celular do hospedeiro. Nos vírus envelopados, as estruturas de ligação geralmente se apresentam sob a forma de espículas, como nos Paramyxovírus e nos vírus sem envelope.
A ligação célula-vírus geralmente está relacionada a um ou grupo de polipeptídeos estruturais, com acontece nos Papilomavírus.
A presença ou ausência de receptores celulares determina o tropismo viral, ou seja, o tipo de célula em que são capazes de ser replicados. Para haver a adsorção, é necessária uma ponte entre as proteínas mediadas por íons livres de cálcio e magnésio, uma vez que as proteínas apresentam carga negativa. Outros fatores vão influenciar diretamente na adsorção do vírus na membrana celular, tais como, temperatura, pH e envoltórios com glicoproteínas.
2) Penetração
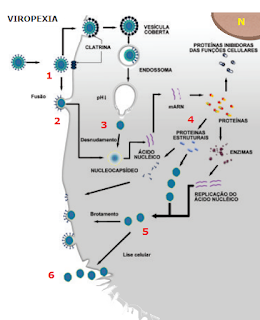
É entrada do vírus na célula. Esta pode ser feita de duas maneiras: fusão e viropexia. A fusão é quando membrana celular e o envelope do vírus se fundem, permitindo a entrada deste no citosol da célula. No caso da família Paramixoviridae, a proteína LL catalisa ligação da membrana com o envelope.
Já a viropexia é uma invaginação da membrana celular mediada por receptores e por proteínas, denominadas clatrinas, que revestem a membrana internamente. Nos dois mecanismos existe uma dependência em relação à temperatura adequada, que fica em torno de 37°C, em vírus que replicam em células de vertebrado.
3) Desnudamento
Neste processo, o capsídeo é removido pela ação de enzimas celulares existentes nos lisossomos, expondo o genoma viral. Além disso, se observa a fase de eclipse, onde não há aumento do número de partículas infecciosas na célula hospedeira. De uma maneira geral, o vírus que possui como ácido nucleico o DNA faz síntese no núcleo, com exceção do Poxvírus, uma vez que precisa da enzima polimerase, encontrada no núcleo da célula. O vírus que possui como genoma o RNA faz a síntese viral no citoplasma, com exceção do vírus Influenza, pois já possui a enzima polimerase.
4) Síntese viral
A síntese viral compreende a a formação das proteínas estruturais e não estruturais a partir dos processos de transcrição (o processo de formação do RNA mensageiro a partir do DNA) e tradução (o processo de converssão de uma molécula, ou sequência nucleotídica, em aminoácidos, formando uma proteína.
Os vírus foram agrupados em sete classes propostas por Baltimore em 1971, de acordo com as características do ácido nucleico e as estratégias de replicação. Nos vírus inseridos nas classes I, III, IV e V, o processo de tradução do RNA mensageiro ocorre no citoplasma da célula hospedeira. Já nos vírus da classe II, este processo ocorre no núcleo. Em todas estas classes, O RNA mensageiro sintetizado vai se ligar aos ribossomos, codificando a síntese das proteínas virais. As primeiras proteínas a serem sintetizadas são chamadas de estruturais, pois vão formar a partícula viral. As tardias são as proteínas não estruturais, que participam do processo de replicação viral. Na classe VI, os vírus de RNA realizam a transcrição reversa formando o DNA complementar (RNA' -›DNA'-›RNA), devido a presença da enzima transcriptase reversa (família Retroviridae). Os vírus da classe VII apresentam um RNA intermediário de fita simples, maior do que o DNA de cadeia dupla que o originou (DNA' -›RNA' -›DNA).
Resumindo, abaixo estão descritas as características principais de cada
Classe I: Ocorre no citoplasma, independente do genoma celular, que é bloqueado.
Classe II: É realizada no núcleo, simultaneamente à síntese do genoma celular.
Classe III: Processa-se no citoplasma; sendo, no início, apenas umas das fitas do ácido nucleico copiada.
Classe IV: Ocorre no citoplasma, por meio de um processo complexo, ainda pouco esclarecido.
Classe V: A fita simples de RNA serve de molde para a formação de genoma viral e sintese de RNA mensageiro.
Classe Vl: Pertence a essa classe a família Retroviridae, que possui uma enzima chamada Transcriptase Reversa, responsável pela síntese de DNA a partir de RNA.
Classe VII: Tem como exemplo a família Hepadnaviridae, cuja característica principal é a formação de um RNA intermediário.
5) Montagem e Maturação
Nessa fase, as proteínas vão se agregando ao genoma, formando o nucleocapsídeo. Alguns vírus, como o Rotavírus, apresentam mais de um capsídeo. A maturação consiste na formação das partículas virais completas, ou vírions, que, em alguns casos, requerem a obtenção do envoltório lipídico ou envelope. Este processo, dependente de enzimas tanto do vírus quanto da célula hospedeira, podendo ocorrer no citoplasma ou no núcleo da célula. De uma forma geral, os vírus que possuem genoma constituído de DNA condensam as suas partes no núcleo, enquanto os de RNA, no citoplasma.
6) Liberação
A saída do vírus da célula pode ocorrer por lise celular ou brotamento. Na lise cellar (ciclo lítico), a quantidade de vírus produzida no interior da célula é tão grande que a célula se rompe, liberando novas partículas virais que vão entrar em outras células. Geralmente, os vírus não envelopados realizam esta ciclo ao passo que os envelopados saem da célula por brotamento. Neste caso, os nucleocapsídeos migram para a face interna da membrana celular e saem por brotamento, levando parte da membrana.
Eventos finais da replicação viral:
1. Transporte do genoma (DNA ou RNA) para o
sítio de processamento (núcleo ou citosol)
2. Transcrição (síntese de mRNA)
3. Síntese de proteínas não estruturais
4. Replicação do material genético
5. Síntese de proteínas estruturais
6. Montagem dos nucleocapsídeos
7. Vesícula com glicoproteínas direcionadas ao complexo de Golgi
8. Transporte das proteínas de envelope à membrana plasmática
9. Liberação de partículas virais por lise (vírus não envelopados), ou por brotamento (vírus envelopados)(WP).
As principais diferenças entre provírus e retrovírus são as seguintes:
Provírus
É um genoma viral integrado ao genoma do hospedeiro.
É uma fase do ciclo (replicação) do vírus.
Eles não contêm enzima transcriptase reversa.
Retrovírus
É um vírus de RNA que tem a capacidade de sintetizar cDNA a partir de RNA pelo processo de transcrição reversa.
Eles são vírus de RNA de fita simples.
Eles contêm uma enzima conhecida como transcriptase reversa.
Visto que a transcriptase reversa não tem a capacidade de revisar o produto polimerizado, o processo de transcrição reversa está muito sujeito a erros.
Nosso corpo possui um tipo específico de transcriptase reversa conhecida como telomerase, responsável pela síntese do DNA telomérico.
os retrovírus são caracterizados pela presença da transcriptase reversa da enzima e do genoma do RNA. Eles produzem dsDNA a partir de ssRNA com a ajuda da transcriptase reversa da enzima e, em seguida, integram seu genoma no genoma do hospedeiro. Em seguida, o genoma retroviral integrado (provírus) transcreve e traduz com as partículas virais (ciclo do retrovírus).
a transcriptase reversa se liga à extremidade 5 'do molde de RNA. Ele sintetiza DNA na direção 5 '-> 3'. Mas lê o modelo de RNA na direção 3 '-> 5'.
Sim, ele pode sintetizar DNA a partir de mRNA. Especificamente, a transcriptase reversa é freqüentemente usada para construir bibliotecas de cDNA a partir de modelos de mRNA.
Os vírus que contêm RNA como genoma são conhecidos como retrovírus.
espumavírus (uma classe de retrovírus) não causam doenças. Nem todas as infecções retrovirais são letais.
Assim pega:
Sexo vaginal sem camisinha;
Sexo anal sem camisinha;
Sexo oral sem camisinha;
Uso de seringa por mais de uma pessoa;
Transfusão de sangue contaminado;
Da mãe infectada para seu filho durante a gravidez, no parto e na amamentação;
Instrumentos que furam ou cortam não esterilizados.
Assim não pega:
Sexo desde que se use corretamente a camisinha;
Masturbação a dois;
Beijo no rosto ou na boca;
Suor e lágrima;
Picada de inseto;
Aperto de mão ou abraço;
Sabonete/toalha/lençóis;
Talheres/copos;
Assento de ônibus;
Piscina;
Banheiro;
Doação de sangue;
Pelo ar.
Glossário dos vírus
AAV - Vírus adenoassociados
cDNA – DNA Complementar
DNA - Ácido desoxirribonucleico
dsDNA - Vírus de DNA cadeia dupla
dsRNA - Vírus RNA cadeia dupla
ssDNA - Vírus de DNA cadeia simples
ssRNA - Vírus RNA cadeia simples
ICNV - Internacional Committee on Nomenclature of Viruses
ICTV - Internacional Committee on Taxonomy of Viruses
MoMuLV - Vírus da leucemia murina de Moloney
RNA - Ácido ribonucleico
mRNA – RNA mensageiro
S3H - Helicases da superfamília 3
SV40 - Simian vírus 40
VIH - Vírus da imunodeficiência humana
Bibliografia
https://edisciplinas.usp.br/pluginfile.php/4397896/mod_resource/content/1/Aula%204.%20Retrovirus.pdf
Leituras recomendadas
CONTÁGIO (Como surgem as pandemias):





































0 comentários:
Postar um comentário